t-SNE 多様体学習
高次元データの各データポイントの類似度を距離として計算し、2次元(または3次元)空間に配置したランダムな点に距離を元にして配置していく。
2次元または多くても3次元に圧縮するのが一般的らしい。また、学習モデルを別の行列に対してtransformすることは出来ないみたい。
分析データ
load_digitsデータを使用します。
from sklearn.datasets import load_digits import mglearn import matplotlib.pyplot as plt import numpy as np from sklearn.model_selection import train_test_split digits = load_digits() fig, axes = plt.subplots(2, 5, figsize=(10,5), subplot_kw={"xticks":(), "yticks":()}) for ax, img in zip(axes.ravel(), digits.images): ax.imshow(img)

PCAデータでの次元削減
比較する為、PCAで削減した成分を可視化。
from sklearn.decomposition import PCA pca = PCA(n_components=2) pca.fit(digits.data) digits_pca = pca.transform(digits.data) colors = ["#476A2A", "#7851B8", "#BD3430", "#4A2D4E", "#875525", "#A83683", "#4E655E", "#853541", "#3A3120", "#535D8E"] plt.figure(figsize=(10,10)) plt.xlim(digits_pca[:,0].min(), digits_pca[:,0].max()) plt.ylim(digits_pca[:,1].min(), digits_pca[:,1].max()) for i in range(len(digits.data)): plt.text(digits_pca[i, 0], digits_pca[i, 1], str(digits.target[i]), color=colors[digits.target[i]], fontdict={"weight":"bold", "size":9}) plt.xlabel("First principal component") plt.ylabel("Second principal component")

比較的分類されていますが、座標(0, 0)付近など重なりが目立ちます。
t-SNEでの次元削減
from sklearn.manifold import TSNE tsne = TSNE(random_state=42) digits_tsne = tsne.fit_transform(digits.data) plt.figure(figsize=(10,10)) plt.xlim(digits_tsne[:,0].min(), digits_tsne[:,0].max()+1) plt.ylim(digits_tsne[:,1].min(), digits_tsne[:,1].max()+1) for i in range(len(digits.data)): plt.text(digits_tsne[i, 0], digits_tsne[i, 1], str(digits.target[i]), color=colors[digits.target[i]], fontdict={"weight":"bold", "size":9}) plt.xlabel("t-SNE feature 0") plt.ylabel("t-SNE feature 1")
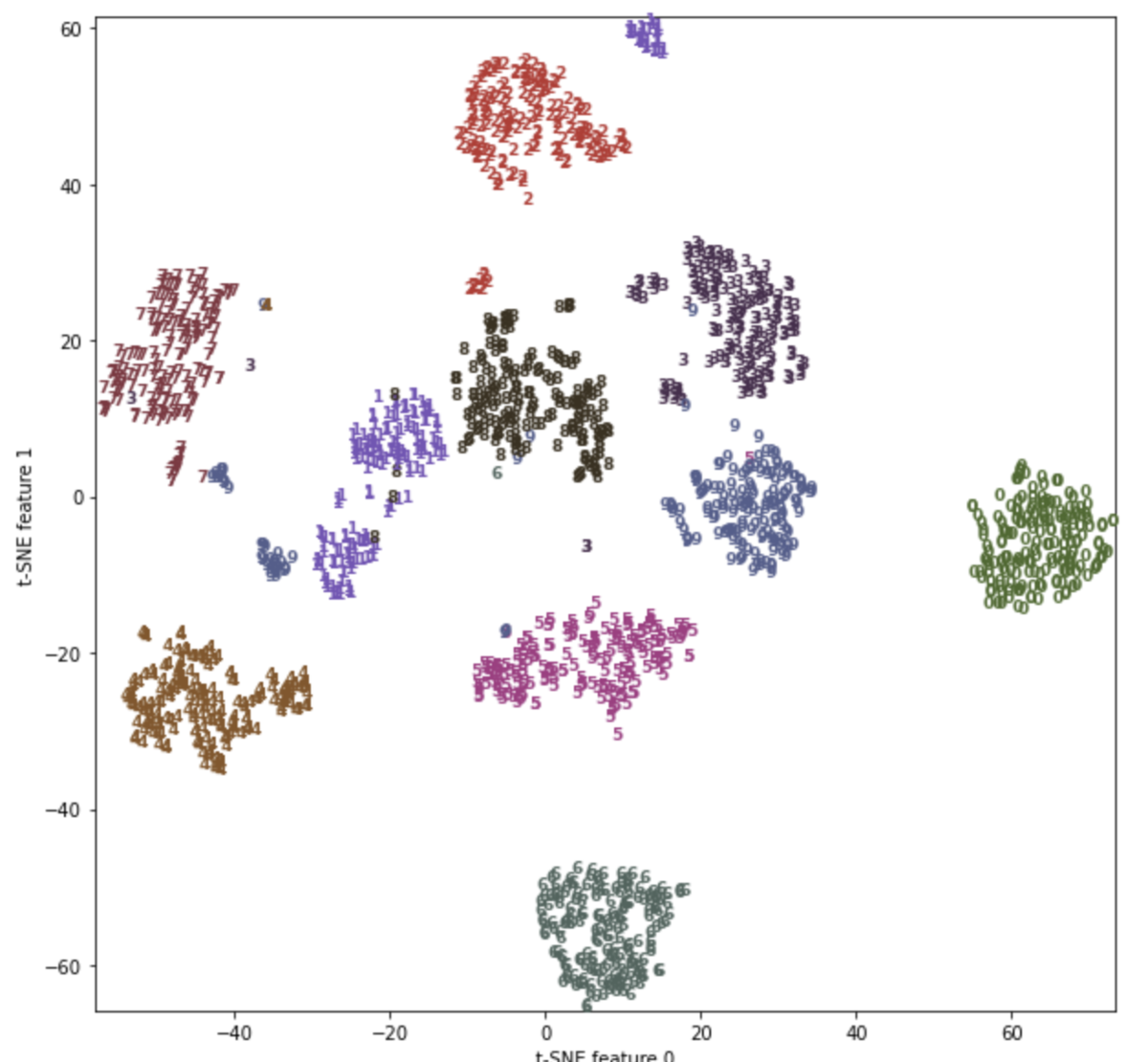
このようにPCAと比べて境界がはっきり分かれた形で可視化することができます。
リンク

